Estructura molecular y mecanismos de acción de los antibióticos
Introducción
[editar]Los antibióticos (ATB) son compuestos orgánicos naturales, semisintéticos o sintéticos que tienen una toxicidad selectiva sobre las bacterias, generalmente a una concentración reducida que el hospedador puede tolerar sin ser dañado [1]. Su descubrimiento, comercialización y aplicación en la práctica clínica permitió el tratamiento de infecciones que en el pasado eran incurables e intervenciones médicas complejas, como el trasplante de órganos y el manejo de pacientes con cáncer, entre otros [2]. Desde que en 1928 Alexander Fleming observó la actividad antimicrobiana de la penicilina, se han descubierto, modificado y sintetizado nuevos ATB (Tabla 1). Para que un ATB sea efectivo contra las bacterias se deben tener tres condiciones:
- La bacteria debe tener un blanco susceptible al ATB.
- El ATB debe alcanzar al blanco en suficiente cantidad.
- La estructura química del ATB no debe ser modificada.
Tabla 1. Cronología del descubrimiento e introducción en la terapéutica de los principales antimicrobianos[3].
[editar]| Año | Evento | Año | Evento |
|---|---|---|---|
| 1929 | Descubrimiento (D.) de la penicilina | 1959 | I. nitroimidazoles |
| 1932 | D. del prontosil (sulfonamidas) | 1960 | I. meticilina |
| 1939 | Aislamiento de la penicilina | 1961 | I. ampicilina |
| 1942 | Introducción (I.) de la penicilina | 1962 | I. ácido nalidíxico |
| 1943 | D. estreptomicina (aminoglucósidos) | 1963 | D. gentamicina |
| 1943 | D. bacitracina | 1964 | I. cefalosporinas |
| 1945 | D. cefalosporinas | 1970 | I. trimetoprim |
| 1947 | D. cloranfenicol | 1972 | I. minociclina |
| 1948 | D. clortetraciclina | 1980 | I. norfloxacina (fluoroquinolonas) |
| 1952 | D. eritromicina | 1993 | I. azitromicina y claritromicina |
| 1956 | D. vancomicina | 2000 | I. linezolid (oxazolidinonas) |
| 1957 | D. rifampicina | 2003 | I. daptomicina (lipopéptidos) |
Estructura de los antibióticos
[editar]Los ATB pueden ser clasificados de varias formas, ya sea por su origen, su efecto sobre las bacterias, o su actividad farmacológica, pero también se les agrupa por su estructura química, ya que de ésta depende la forma en que actúan sobre la bacteria, y contra qué tipos de bacterias pueden actuar.
ß-lactámicos
[editar]
Este grupo de antibióticos es el más grande y prescrito para el tratamiento de infecciones bacterianas, debido a su efectividad y a que presentan menos efectos adversos en comparación con otros antibióticos.
En su estructura química, todos presentan un anillo betalactámico, que consiste en un anillo heterocíclico de tres átomos de carbono y uno de nitrógeno. Las cadenas laterales determinan la actividad antimicrobiana y su farmacodinámica.
Los ß-lactámicos bloquean la transpeptidación del peptidoglicano al unirse a las PBP (penicillin-binding proteins), debido a la semejanza estructural entre estos antibióticos y los precursores pentapeptídicos del peptidoglicano. y las PBP elaboran un complejo antibiótico-enzima que destruye el peptidoglicano y activa la autolisina bacteriana endógena, por lo que se provoca la lisis osmótica de la bacteria. Debido a que las penicilinas actúan sobre la síntesis activa, sólo afecta a las células en crecimiento activo.
Penicilinas
[editar]
Presentan un anillo 6-aminopenicilánico, que consiste en el anillo betalactámico unido a un anillo tiazolidínico y sus propiedades dependen de la cadena unida a su grupo amino.
Cefalosporinas
[editar]
Tienen un ácido-7-cefalosporánico unido al anillo betalactámico . Se han modificado las cadenas laterales para aumentar su espectro antibacteriano hacia bacterias gramnegativas. A estos cambios se les han denominado generaciones de cefalosporinas.
Monobactámicos
[editar]El Aztreonam es el único antibiótico de este grupo utilizado en medicina, que presenta un anillo betalactámico unido a un ácido sulfámico y una treonina , lo que le da mayor estabilidad ante las β-lactamasas.
Carbapenemes
[editar]
Su estructura difiere en gran medida de los otros betalactámicos, porque en vez de un átomo de azufre, presenta en su anillo un átomo de carbono en la posición 1, de ahí su nombre. Presentan el mayor espectro de actividad, y son resistentes a las β-lactamasas.
Glucopéptidos
[editar]Los glucopéptidos (vancomicina y teicoplanina) impiden la transferencia del disacárido pentapéptido al aceptor de la pared celular, porque estos compuestos recubren el extremo D-alanina-D-alanina del disacárido-pentapéptido precursor, evitando la elongación del peptidoglicano, y también alteran la permeabilidad de la membrana citoplasmática y la síntesis de ARN, Sus múltiples mecanismos de acción contribuyen a la baja frecuencia de desarrollo de resistencia.
Mecanismos de Acción
[editar]Se entiende por mecanismo de acción las funciones que llevan a cabo los ATB para presentar un efecto sobre el organismo. Estos van desde la modificación de una célula u órgano diana, hasta la inhibición de ciertos sustratos e incluso modificaciones a nivel celular. A continuación, mencionamos algunos de los principales mecanismos de acción propios de los ATB.
Inhibidores de la fase de transporte de precursores
[editar]● Bacitracina

La bacitracina se une al transportador lipídico de los polímeros lineales del complejo NAG-NAM pentapéptido a través de la membrana citoplásmica, hasta la pared en formación. Este antibiótico es activo contra cocos grampositivos (excepto estreptococos), pero debido a la elevada nefrotoxicidad, sólo se utiliza de forma tópica.
Inhibidores de la síntesis de precursores
[editar]● Fosfomicina

La fosfomicina inhibe la piruviltransferasa, enzima causante de la adición del fosfoenolpiruvato a la molécula de UDP-NAG para formar el precursor UDP-NAM. La fosfomicina es un análogo estructural del fosfoenolpiruvato y se une covalentemente con la enzima. Debido a su pequeño tamaño, puede atravesar la barrera de peptidoglicano y finalmente atraviesa la membrana citoplásmica a través de sistemas de transporte activo, que se favorece en presencia de glucosa-6-fosfato. Es un antibiótico de amplio espectro que incluye bacilos gramnegativos y grampositivos y Staphylococcus spp. (excepto S. saprophyticus y S. capitis).
● Cicloserina

La cicloserina inhibe la actuación de la enzima que convierte la L-ala a D-ala, así como la reacción de unión de dos D-ala por su estructura similar al aminoácido D-alanina y al que suplanta en la síntesis del peptidoglicano. Se usa como fármaco de segunda línea contra Mycobacterium tuberculosis debido a la elevada toxicidad, siendo preponderantes las neuropsiquiátricas.
Alteración de la membrana celular
[editar]● Polimixina B y Colistina
Las polimixinas pueden unirse a varios ligandos de células del tejido corporal y son tóxicas para los riñones y el sistema nervioso. Otro antibiótico que actúa en la membrana es la gramidicina, la cual produce desacoplamiento de la fosforilación oxidativa y la formación de poros por donde puede haber pérdida del contenido citoplasmático de la bacteria. Son activos exclusivamente frente a bacilos gramnegativos aerobios, incluidos P. aeruginosa y A. baumannii multirresistentes. No son activos frente a microorganismos anaerobios.
Alteración o inhibición de la síntesis de proteínas
[editar]● Aminoglucósidos

Los aminoglucósidos comprenden un gran grupo de compuestos semisintéticos o naturales, (estreptomicina, neomicina, kanamicina, amikacina, tobramicina, gentamicina, espectinomicina, paromomicina) que tienen amino azúcares ligados glucosídicamente a los aminociclitoles, alcoholes cíclicos que también están sustituidos con grupos funcionales amino.
La mayoría son agentes bactericidas y comparten el mismo rango general de actividad antibacteriana, comportamiento farmacocinético, y una tendencia a causar daño renal. El primer paso es la unión del aminoglucósido a una proteína receptora específica (P12 en el caso de la estreptomicina) en la subunidad 30S del ribosoma microbiano, se bloquea la actividad normal del complejo de iniciación y el mensaje del mRNA se lee mal en la región de reconocimiento del ribosoma, provocando la formación de una proteína no funcional, y causan la muerte de las bacterias.
● Macrólidos
Los macrólidos (eritromicina, oleandomicina), están compuestos por un anillo lactónico macrocíclico, que puede tener 14, 15 o 16 átomos de carbono, al que se unen diversos desoxiazúcares . Al unirse a la subunidad 50S interfieren con la actividad de la peptidil transferasa, con la translocación o con ambas funciones.
● Tetraciclinas

Contienen un anillo de naftaleno de cuatro átomos y químicamente son derivados de la naftacenocarboxamida policíclica, núcleo tetracíclico, de donde deriva el nombre del grupo. Actúan en la subunidad ribosomal 30S inhibiendo la unión del aminoacil tRNA al ribosoma, y de esta manera, impiden la introducción de nuevos aminoácidos en la cadena nueva de péptidos, pero la unión es temporal, y sólo ejerce un efecto bacteriostático.
● Gliciclinas

La tigeciclina es el primer miembro de esta familia, con su estructura modificada a partir de la minociclina (una tetraciclina), e inhiben la síntesis de proteínas de manera similar a las tetraciclinas; sin embargo, son bactericidas por su unión más ávida con el ribosoma. La tigeciclina es activa contra una gran variedad de bacterias, tanto grampositivas como gramnegativas, incluidas algunas cepas que son resistentes a las tetraciclinas, y tiene gran actividad frente a bacterias multirresistentes.
● Clorafenicol

El cloranfenicol se une a la subunidad 50S del ribosoma. Interfiere con el enlace de nuevos aminoácidos en la formación de la cadena peptídica, porque el cloranfenicol inhibe a la peptidiltransferasa. El cloranfenicol es bacteriostático, y la proliferación de los microorganismos se restablece cuando el fármaco se suspende.
● Lincosamidas
Son una clase de antibióticos que incluyen a la clindamicina y la lincomicina , y se unen a la porción 23s de la subunidad 50S del ribosoma bacteriano, de manera similar a los macrólidos, y son similares también en cuanto al sitio de unión, su actividad antibacteriana y el modo de acción.
Inhibición de síntesis de ácidos nucleicos
[editar]
La rifampicina inhibe la actividad de la RNA polimerasa bacteriana dependiente de DNA, uniéndose en forma firme a esta enzima. La RNA polimerasa es una enzima que se une a un factor que confiere especificidad para el reconocimiento de los sitios requeridos para iniciar la transcripción del DNA. La rifampicina se une a subunidades de la RNA polimerasa e interfiere específicamente con la iniciación del proceso, pero no tiene efecto cuando la polimerización se ha iniciado.
Otro mecanismo para inhibir la replicación del DNA se produce por antimicrobianos que inhiben la actividad de la DNA girasa, involucrada en el rompimiento y reunión de tiras de DNA. La girasa está constituida por dos componentes, A y B. El ácido nalidíxico y sus derivados, las fluoroquinolonas, se unen al componente A de la DNA girasa e inhibe su acción. El ácido nalidíxico tiene acción antimicrobiana sólo contra especies gramnegativas. La subunidad B de la DNA girasa puede ser inhibida por agentes como la novobiocina, de uso restringido debido a su toxicidad.
Para muchos microorganismos, el ácido p-aminobenzoico (PABA) es un metabolito indispensable para la síntesis de ácido fólico, precursor importante para la síntesis de ácidos nucleicos. Las sulfonamidas con análogos estructurales de PABA e inhiben a la dihidropteroato sintetasa, entran en la reacción en lugar del PABA y forman análogos no funcionales de ácido fólico, lo que impide la proliferación de la célula bacteriana. El trimetoprim inhibe al ácido dihidrofólico reductasa con una eficacia 50 000 veces mayor en las bacterias que en las células de mamífero. Esta enzima reduce al ácido dihidrofólico para formar ácido tetrahidrofólico, una fase en la secuencia que provoca la síntesis de purinas y finalmente DNA.
Inhibición de Betalactamasas
[editar]
Los inhibidores de las betalactamasas (Figura 19.1) carecen de actividad antimicrobiana, por lo que se administran con antibióticos betalactámicos, pero actúan de manera reversible o irreversible al inhibir a muchas de las enzimas betalactamasas, reduciendo la resistencia a ciertas bacterias en contra de estos antibióticos. Los únicos inhibidores disponibles para uso clínico son el ácido clavulánico (Fig. 19), el tazobactam y el sulbactam.
Mecanismos de Resistencia
[editar]La resistencia a los ATB se puede definir teniendo en cuenta los criterios farmacocinéticos y farmacodinámicos para determinar valores por encima de los cuales es difícil obtener una concentración terapéuticamente útil. Si la concentración inhibitoria mínima (MIC) para una bacteria está por encima de esos valores de concentración, existe el riesgo de que la infección no se pueda tratar con éxito. Por lo tanto, los microorganismos se clasifican como resistentes cuando sus MIC están por encima de un umbral predefinido. La resistencia a los efectos de los ATB para el tratamiento de enfermedades es resultado de un consumo poco moderado e inconsciente. Los ATB se encuentran entre las drogas utilizadas con mayor frecuencia en la comunidad, representando así alrededor del 12% del total de prescripciones a nivel mundial. El serio problema global del aumento de resistencia bacteriana es una consecuencia del uso y abuso de los ATB tanto a nivel hospitalario como a nivel de comunidad. Los mecanismos de resistencia bacteriana son modificaciones mecánicas o estructurales que adquieren las bacterias patógenas desencadenando una disminución o incluso anulación total en la respuesta frente a los ATB y se pueden clasificar de la siguiente manera[4]:
Transmisión horizontal y vertical de genes de resistencia
[editar]Las cepas de bacterias que transportan mutaciones cromosómicas que confieren resistencia sobreviven y crecen bajo la presión selectiva del uso de antibióticos, en cambio, mata las cepas susceptibles. Se da por lo tanto una propagación de genotipos resistentes. Este tipo de selección se denomina evolución vertical porque los elementos genéticos asociados a la resistencia se transmiten de una célula a otra a través de la duplicación celular. Las bacterias también desarrollan resistencia a través de la adquisición de nuevo material genético de organismos resistentes. Este tipo de selección se denomina evolución horizontal. Los mecanismos de intercambio genético incluyen eventos como la conjugación, la transducción y la transformación. Durante la conjugación se transfiere un plásmido o DNA genómico que contiene genes de resistencia a una bacteria receptora a través de un puente de acoplamiento. Durante la transducción, los genes de resistencia se transfieren a través de bacteriófagos. Finalmente, las llamadas bacterias competentes pueden adquirir e incorporar genes de resistencia de otras bacterias que han liberado su ADN al medio ambiente después de la lisis celular, por transformación. [5]

Resistencia bacteriana a antibióticos por degradación enzimática o modificación química
[editar]Los mecanismos de activación antibiótica comparten muchas similitudes con reacciones enzimáticas bien caracterizadas involucradas en el metabolismo primario. Las enzimas que confieren resistencia mediante la destrucción o modificación de antibióticos utilizan un conjunto de estrategias químicas que pueden agruparse funcionalmente en hidrólisis, transferencia grupal y mecanismos redox.
- Hidrólisis: Varios tipos de enzimas confieren resistencia al apuntar y escindir enlaces químicos que son propensos a la hidrólisis. Estas enzimas requieren agua para la catálisis y son excretadas por bacterias, por lo que interceptan los antibióticos antes de que entren en contacto con su objetivo bacteriano.
- β-Lactamasas: Hay dos clases principales de β-lactamasas basadas en el mecanismo molecular de hidrólisis del anillo de β-lactama: (i) Ser-β-lactamasas, como BlaZ, que funcionan a través de la acción de un sitio activo de nucleófilos Ser y (ii ) metalolactamasas que activan el agua a través de un centro Zn2 +.
- Esterasas (macrólidos): Los macrólidos son ciclados por una tioesterasa. Este enlace está dirigido por la enzima de resistencia a macrólidos que funciona en modo de apertura inversa del anillo.
- Epoxidasas : La resistencia enzimática a este antibiótico ocurre mediante la destrucción del epóxido reactivo de la apertura del anillo.
- Transferasas antibióticas: Las transferasas representan la familia más grande de enzimas de resistencia. Estas enzimas modifican covalentemente los antibióticos, perjudicando su unión a la diana. Sus actividades incluyen O-y N-acilación, O-fosforilación, O-nucleotidilación, O-ribosilación, O-glicosilación y transferencia de tiol. Todas estas reacciones requieren un cosustrato, incluyendo adenosina trifosfato (ATP), acetil-CoA, nicotinamida adenina dinucleótido (NADH) uridina difosfato (UDP) glucosa o glutatión, y, en consecuencia, todas estas enzimas solo funcionan en el citosol.
- Acetiltransferasas: Tienen como diana el hidroxilo (para O-acetilación) y / o grupos amina (para N-acetilación) en antibióticos. El éster o amida resultante es biológicamente estable y esencialmente irreversible sin la acción de una esterasa o amidasa afines. Pueden ser Aminoglucósido o cloranfenicol transferasas
- Fosfotransferasas. Pueden ser aminoglucósido o macrólido transferasas. Fosforilan los agentes antimicrobianos, es decir, añade un grupo fosfato.
- Nucleotidil-transferasas: Transfieren la fracción de nucleótido monofosfato de NTP a un grupo hidroxilo aceptante en el antibiótico.
- La ADP-ribosilación del inhibidor de la ARN polimerasa rifampicina (rifampicina), utilizada en el tratamiento de infecciones causadas por Mycobacterium tuberculosis, es hasta ahora el único ejemplo bien documentado de este tipo de modificación en la resistencia a los antibióticos. En las micobacterias, una rifampicina única ADP-ribosiltransferasa (ARR) interfiere con la actividad de este producto.
- Glicosiltransferasas: Infrecuente en patógenos, se cataliza la glucosilación de la eritromicina y otros macrólidos en la posición 2 'del azúcar desosamina usando glucosa UDP como donante de glucosa.
- Enzimas Redox: La oxidación o reducción de antibióticos no ha sido explotada con frecuencia por bacterias patógenas. El ejemplo mejor estudiado de esta estrategia es la oxidación de antibióticos de tetraciclina por TetX, una enzima que cataliza la monohidroxilación de antibióticos de tetraciclina dependiente del oxígeno.
Consecuencias de la resistencia bacteriana
[editar]Como ya se mencionó, la resistencia bacteriana puede suceder de manera independiente al consumo de ATB, sin embargo debido al consumo irregulado de los mismos ha dado lugar a una nueva generación de cepas bacterias resistentes a la mayoría de ATB derivando eventualmente en una crisis de salud pública, en especial si tenemos en cuenta que las bacterias tiene la capacidad de generar resistencia de huésped a huésped y que además no solo afectan a humanos sino a una amplia gama de animales no humanos provocando que cada vez se tenga un menor control bactericida sobre las cepas bacterianas. De acuerdo con el incremento en la resistencia bacteriana a los ABS hasta hoy conocidos, resulta necesario la creación de nuevos ABS multirresistentes, además de adquirir conciencia sobre el uso racional y adecuado de ABS como sociedad.
Antibióticos de nueva generación
[editar]Debido a los diversos reportes desde el año 2014 de la Organización Mundial de la Salud (OMS), sobre la capacidad de los patógenos bacterianos a adaptarse a las distintas condiciones en las que se encuentren, y el uso irresponsable que se le ha dado a los diversos ATB, ha provocado que se genere una resistencia a estos medicamentos ya conocidos, amenazando a la salud mundial; en respuesta a estos reportes se está trabajado en el desarrollado nuevos ATB, los cuales se describen a continuación.[6]
Estreptograminas
[editar]Es un grupo de ATB formado por dos componentes, la estreptogramina a y estreptogramina b, siendo la estreptogramina a conocida como D por Dalfopristin, y la estreptogramina b conocida como Q por Quinupristin este último es el componente B, y Dalfopristin es el componente A. ambos tienen en su estructura un anillo de macrolactona.
Su mecanismo de acción es a nivel del ribosoma bacteriano, donde el componente A se une al peptidil tRNA y bloquea la unión de nuevos aminoácidos, a continuación el componente B impide la elongación de la cadena peptídica, el componente A aumenta la afinidad del componente B por la subunidad 50S, dando la combinación de A+B,que es un bactericida.
Su espectro antimicrobiano es efectivo con la mayoría de cepas de bacterias gram positivas (menos E. faecalis) y algunas gram negativas como H. influenzae, Neisseria spp., M. catarrhalis, sin embargo no presenta actividad antimicrobiana frente a enterobacterias, Pseudomonas y Acinetobacter spp

Oxazolidinonas
[editar]Estos compuestos interfieren con la traducción al impedir la formación de N-formilmetionil-tRNA, que es el complejo de iniciación en el ribosoma 23S. El más representativo es el linezolid, este inhibe la síntesis proteica uniéndose a la subunidad 50S ribosomal, donde compite en este sitio de unión con el cloranfenicol y con lincosamidas, no interfiere con la elongación de la cadena peptídica y con la traslación, sin embargo, ya que el sitio de unión ribosomal está cercano a la interfase de la porción 30S por lo que logra inhibir el complejo de iniciación.
Daptomicina
[editar]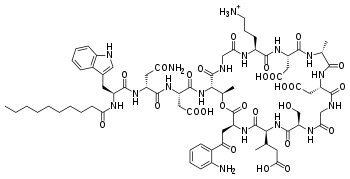
Se trata de un lipopéptido cíclico natural, que es activo únicamente contra las bacterias gram-positivas. Su mecanismo de acción consiste en la unión (en presencia de iones de calcio) a las membranas bacterianas de las células tanto en fase de crecimiento como estacionaria, causando una despolarización y conduciendo a una rápida inhibición de la síntesis de proteínas, de ADN y de ARN. Dando como resultado la muerte de la célula bacteriana con una lisis celular.
Teixobactina
[editar]
La Teixobactina un nuevo ATB que aún está en etapa de pruebas, un equipo de Northeastern University / Bonn / Novobiotic (y Selcia) publicó un artículo en la revista Nature sobre este nuevo ATB, su mecanismo resulta en que se une a algunos de los precursores del peptidoglicano, el lípido II y el lípido III, similar a la vancomicina que se une al lípido II, sin embargo, la Teixobactina tiene un espectro más amplio. Este mecanismo hace que el desarrollo de la resistencia no sea tan sencillo: la presión de selección es más un golpe de rebote que un golpe directo.
Complestatina y Corbomicina
[editar]Son un conjunto de ATB derivados de los glucopéptidos , por lo tanto actúan directamente sobre la pared de las células bacterianas, con la diferencia de que no actúan en los procesos de síntesis de la pared celular, en su lugar, estos ATB fortalecen la pared de la células bacterianas de modo que les hace imposible multiplicarse en el hospedero. El mecanismo de acción consiste en la inhibición de la producción de peptidoglicano (proteína esencial para la producción de la pared celular), (Culp et al. 2020)[7]
Reacciones adversas de antibióticos
[editar]Hoy, del 5% al 15% de los pacientes en países desarrollados tienen una etiqueta de alergia a la penicilina. Si bien las reacciones adversas a antibióticos (ADR) son comúnmente reportadas, la hipersensibilidad mediada inmunológicamente es poco común y la alergia a los antibióticos mediada por IgE verdadera se verifica sólo en una pequeña minoría. Para aquellos con reacciones de hipersensibilidad a antibióticos real, la evaluación de especialidad apropiada está indicada para prevenir la futura morbilidad y mortalidad relacionadas con ADR. A pesar de la amenaza asociada con la verdadera alergia a los antibióticos, la mayor carga recae en aquellos que informan una alergia a la penicilina que no la tienen. Estos pacientes tienen múltiples secuelas negativas de por vida que comienzan con profilaxis y tratamiento de infecciones de espectro amplio inferior e innecesariamente. Esto se debe a que son prescritos antibióticos de más amplio espectro y de menor especificidad. (Blumenthal et al, 2018).
Referencias
[editar]- ↑ Brooks, G. F., Carroll, K. C., Butel, J. S., Morse, S. A., y Mietzner, T. A. (2011). Jawetz, Melnick y Adelberg. Microbiología médica. México, D.F.: McGraw-Hill Interamericana Editores.
- ↑ Murita, J. M., y Arias, C. A. (2016). Mechanisms of Antibiotic Resistance. Microbiol Spectr, 1-37.
- ↑ Belloso, W. H. (2009). Historia de los antibióticos. Rev. Hosp. Ital. B. Aires, 102-120.
- ↑ Gualerzi, C. O., Brandi, L., & Fabbretti, A. (Eds.). (2013). Antibiotics : Targets, mechanisms and resistance.
- ↑ Gualerzi, C. O., Brandi, L., & Fabbretti, A. (Eds.). (2013). Antibiotics : Targets, mechanisms and resistance.
- ↑ Bogdanowicz E. (2011). Nuevos antibióticos para gérmenes resistentes. Recuperado el 20 de Mayo del 2020 de Sociedad Argentina de Pediatría. Sitio web: https://www.sap.org.ar/docs/congresos/2011/infectologia/bogdanowicz_antibioticos.pdf
- ↑ Culp, E.J., Waglechner, N., Wang, W. et al. Evolution-guided discovery of antibiotics that inhibit peptidoglycan remodelling. Nature 578, 582–587 (2020). https://doi.org/10.1038/s41586-020-1990-9
